La UV i el CSIC usen una IA per a identificar quins bacteris superresistents pot atacar un virus
Es tracta d’un gran avanç en la lluita contra la resistència als antibiòtics mitjançant tractaments personalitzats
Un equip d'investigació de l'Institut de Biologia Integrativa de Sistemes (I2SysBio), centre mixt de la Universitat de València i del Consell Superior d'Investigacions Científiques (CSIC), ha desenvolupat un innovador sistema d'intel·ligència artificial per a predir quins bacteris poden ser atacats per virus bacterians (fags) en funció de la seqüència d'un enzim clau: la depolimerasa. L'estudi ha sigut publicat en la revista Nature Communications.
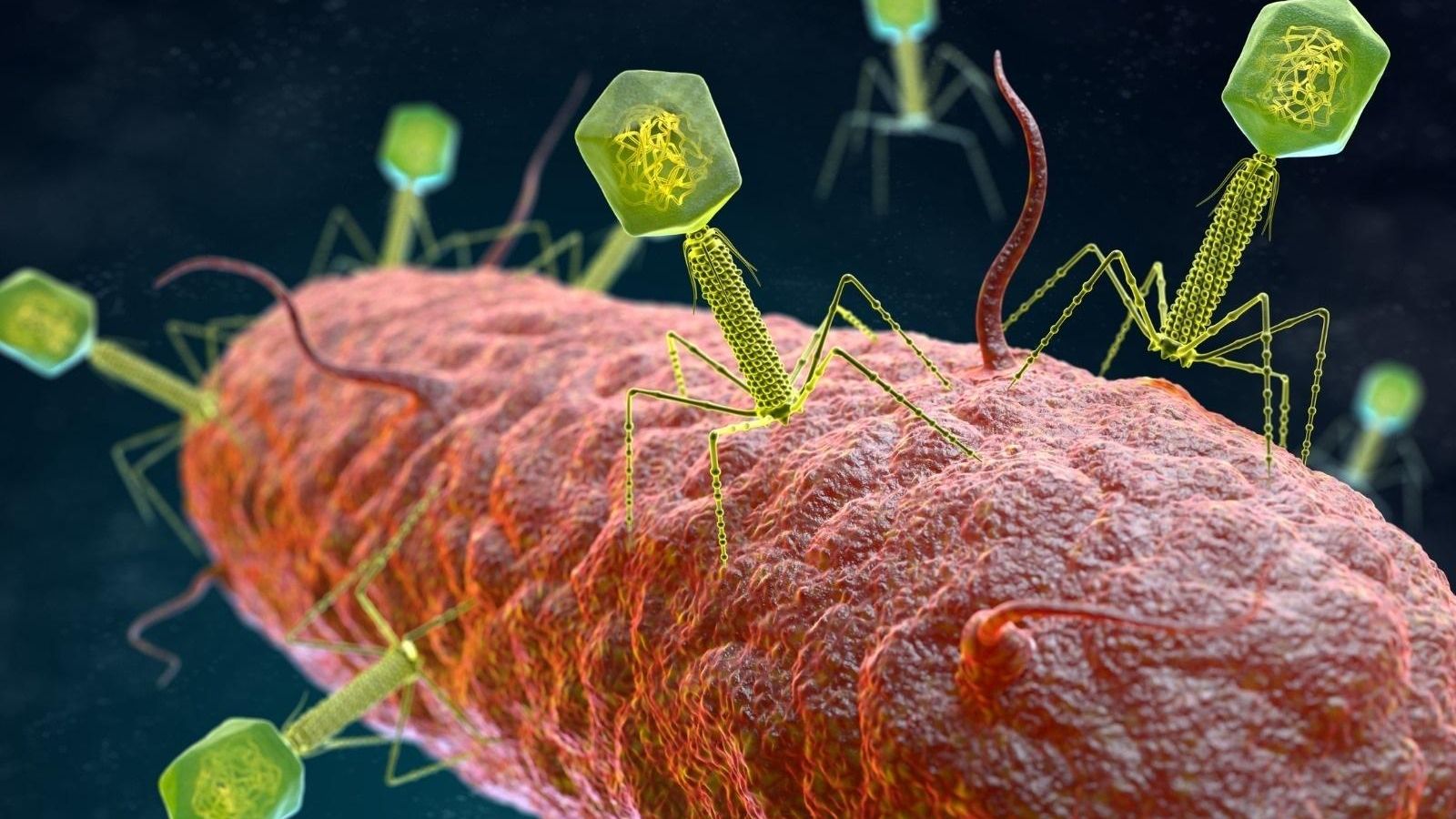
Segons ha informat la institució acadèmica en un comunicat, la resistència als antibiòtics dificulta "cada vegada més el tractament d'infeccions bacterianes". Els fags, que ataquen bacteris, es presenten com a alternativa al tractament antibiòtic normal. No obstant això, identificar quin fag és eficaç enfront de cada bacteri és “complex”. L’estudi proposa l'ús d'intel·ligència artificial per a facilitar este procés de predicció.
Per a l'elaboració del model han utilitzat el bacteri Klebsiella, inclòs en la llista de patògens bacterians “prioritaris” de l'OMS, responsable d'infeccions hospitalàries greus i amb una gran resistència als antibiòtics. Els bacteris Klebsiella estan protegits per càpsules de polisacàrids que impedeixen l'activitat dels antibiòtics, així com l'entrada dels fags.
Per a superar eixa barrera, molts fags produïxen depolimerases, enzims que degraden estes càpsules i permeten l'entrada del bacteriòfag perquè infecte el bacteri i contribuir al seu tractament.
No obstant això, l'enorme diversitat genètica d'estes càpsules —s'han registrat més de 100 serotips d'estes estructures en Klebsiella— ha dificultat la predicció de quin fag pot ser l'indicat per a aconseguir travessar la càpsula i infectar al bacteri. Alhora, esta gran varietat de serotips de càpsules converteix la Klebsiella en un "model ideal" per a estudiar la interacció entre fags i càpsules.
Un “avanç clau” contra els bacteris superresistents
Amb este objectiu, l'equip d'investigació ha desenvolupat una eina pionera que aprofita la informació genètica de milers de bacteris Klebsiella i els virus “adormits” (profags) integrats en el seu genoma. Analitzant més de 74.000 profags i quasi 20.000 seqüències de depolimerases, els investigadors han creat una base de dades que associa cada enzim amb el tipus de càpsula bacteriana que pot degradar.
Mitjançant tècniques avançades d'aprenentatge automàtic i models d'intel·ligència artificial inspirats en el processament del llenguatge natural (similars als que usen els traductors automàtics), han aconseguit predir amb gran precisió el “tropisme” o especificitat de cada depolimerasa, és a dir, quins tipus de càpsula bacteriana pot reconéixer i destruir.
Este estudi aporta un "avanç clau" per a la biotecnologia basada en fags o els seus components, perquè permet predir la seua especificitat. Això és fonamental per a dissenyar futures aplicacions, com a solucions contra el biofilm, l'estructura protectora que formen alguns bacteris per a adherir-se a superfícies i resistir tractaments.
A més, tot i que s'ha utilitzat Klebsiella com a model, esta metodologia pot utilitzar-se contra qualsevol altre bacteri productor de càpsules. Això inclou la majoria dels patògens prioritaris recollits per l'OMS.
En síntesi, l’estudi proposa la resolució de les prediccions d'interacció entre fags i hostes de dos maneres: en primer lloc, aprofitant les dades contingudes en genomes bacterians (profags), cosa que permet obtindre importants dades d'entrenament; i, en segon lloc, proposant una arquitectura que permeta entrenar el model amb totes les espècies bacterianes alhora (de manera integradora).